Die Gene des HLA-Systems kodieren Polypeptidketten auf der Oberfläche der meisten menschlichen Zellen (s. Biochemie). Sie sind auf dem kurzen Arm des Chromosoms 6 lokalisiert und umfassen etwa 1/1000 des gesamten menschlichen Genoms. Da die Polypeptidketten des HLA-Systems bei Abstoßungsreaktionen im Rahmen von Organ- und Gewebetransplantationen eine wichtige Rolle spielen, bezeichnet man diesen Abschnitt des menschlichen Genoms auch als „Major Histocompatiblity Complex“ (MHC-Komplex).
Funktional unterscheidet man im MHC-Komplex 3 Genregionen:
Klasse I-Region
Klasse II-Region
Klasse III-Region
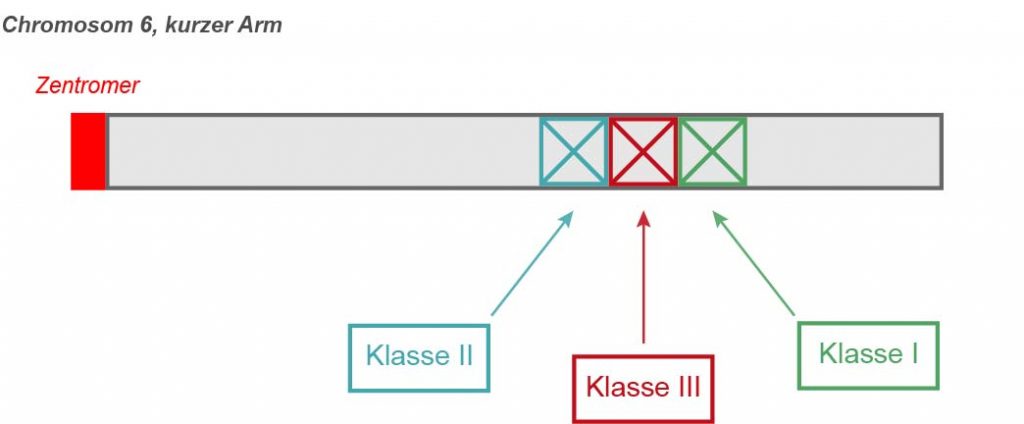
Klasse I-Region
In der Klasse I-Region (Klasse I-Locus) werden unterschiedliche Gengruppen kodiert:
- Klassische HLA-Gene (A, B, C)
- Nicht-klassische HLA-Gene (E, F, G, MICA, MICB, HFE-Hämochromatose-Gen)
- Pseudogene (H, J, K, L, N, P, S, T, U, V, W, X, Y, Z)
Klinisch relevant sind in erster Linie die klassischen HLA-Gene A, B und C. Alle anderen Gene kodieren entweder keine vollständigen Polypeptidketten (Pseudogene) oder solche, welche nur auf wenigen Zellarten exprimiert werden bzw. nur geringe Polymorphismen aufweisen.
Auf den Genorten A, B und C findet sich eine ausgerägte multiple Allelie. So sind inzwischen sowohl für den A- als auch für den B- und C-Locus jeweils mehrere tausend Allele bekannt (und nahezu täglich kommen neue hinzu). Einzelne dieser Allele kodieren sehr ähnliche Polypeptide und werden daher zu Gruppen zusammengefasst, z.B. zur Allelgruppe A1, A30, B7, B8, Cw 3 oder Cw 7 (usw.). Während mit den herkömmlichen serologischen Methoden nur diese Allelgruppen zu erfassen sind, können heute mit modernen molekulargenetischen Methoden auf DNA-Ebene alle einzelnen Allele nachgewiesen werden.
Klasse II-Region
Auch in der Klasse II-Region werden unterschiedliche Gengruppen kodiert:
- DR-Gene (DR-A, DR-B1, DR-B2, DR-B3, DR-B4, DR-B5, DR-B6, DR-B7, DR-B8, DR-B9)
- DQ-Gene (DQ-A1, DQ-A2, DQ-B1, DQ-B2, DQ-B3)
- DP-Gene (DP-A1, DP-A2, DP-A3, DP-B1, DP-B2)
Der DR-Locus umfasst grundsätzlich das Gen DR-A und mehrere Gene DR-B (DR-B1, DR-B2, DR-B3 usw.). Während für das Gen DR-A nur wenige Allele existieren, ist für die DR-B-Gene eine mannigfaltige multiple Allelie bekannt (z.B. wurden für DR-B1 bisher weit über 1000 Allele gefunden).
Auch für den DQ-Locus sind die einzelnen Gene weitgehend bekannt. So wurden dort zwei Gene DQ-A (DQ-A1, DQ-A2) und drei Gene DQ-B (DQ-B1, DQ-B2, DQ-B3) nachgewiesen. Auch für diese Gene existiert eine multiple Allelie. Allerdings sind auf dem DQ-Locus insgesamt weniger Allele bekannt als auf dem DR-Locus.
Ähnlich ist die Situation am DP-Locus. Auch hier gibt es drei DP-A- (DP-A1 DP-A2, DP-A3) und zwei DP-B-Gene (DP-B1, DP-B2), die durch unterschiedliche Allele besetzt sein können.
Neben diesen „klassischen Genen“ finden sich in der Klasse-II-Region weitere sog. „nicht-klassische“ Gengruppen wie die DM-Gene oder die DO-Gene, die ebenfalls durch einzelne Gene mit entsprechenden Allelen gekennzeichnet sind.
Allerdings sind (wie auch im Bereich der Klasse I-Region) nicht alle Gene der Klasse II-Region aktiv und weisen als Genprodukt ein funktionales Polypeptid auf. Viele Gene sind Pseudogene, d.h. sie sind zwar als Gene vorhanden, aber „stumm“, d.h. sie resultieren nicht in einer spezifischen Aminosäuresequenz auf der Zelloberfläche. Hierzu gehören beispielsweise die Gene DR-B2, DR-B6, DR-B7, DR-B8, DR-B9 sowie DP-A2, DP-A3 und DP-B2.
Auch im DR-, DQ- und DP-Bereich können mehrere Allele zu Gruppen zusammengefasst werden. Diese Gruppen entsprechen in etwa – vergleichbar der Klasse I – den mit serologischen Methoden nachweisbaren Klasse II-Entitäten. Eine Auflösung auf Einzel-Allel-Ebene ist nur mittels gentechnologischer Methoden möglich.
Klasse III-Region
In der Klasse III-Region sind Gene lokalisiert, welche die Proteine des Komplementsystems steuern (z.B. C2, C4a, C4b). Darüber hinaus finden sich dort die Gene für die Tumornekrosefaktoren TNF alpha und TNF beta, die Hitzeschockproteine (spielen möglicherweise eine Rolle für die zelluläre Antwort bei unspezifischen Schädigungen wie Hitze) und andere.
Die Vererbung
Die Kombination aller Allele in einem Chromosom bilden einen sog. Haplotyp, der in toto vererbt wird. Der Vererbungsmodus entspricht den klassischen Mendelschen Regeln (kodominante Vererbung).
Beispiel:
(Klasse I: serologische Nomenklatur; Klasse II: molekulargenetische Nomenklatur)
Vater | Mutter | ||||
|---|---|---|---|---|---|
| HLA-A | 2 | 3 | 1 | 29 | Klasse I |
| HLA-B | 8 | 60 | 8 | 35 | Klasse I |
| HLA-C | 7 | 3 | 7 | 4 | Klasse I |
| HLA-DRB1* | 13:01 | 08:01 | 07:01 | 03:01 | Klasse II |
| HLA-DQA1* | 01:03 | 04:01 | 02:01 | 05:01 | Klasse II |
| HLA-DQB1* | 06:03 | 04:02 | 03:03 | 02:01 | Klasse II |
| HLA-DPB1* | 04:01 | 15:01 | 09:01 | 04:01 | Klasse II |
Mögliche Konstellation bei den Kindern
Kind 1 | Kind 2 | Kind 3 | Kind 4 | ||
|---|---|---|---|---|---|
| HLA-A | 2/1 | 2/29 | 3/1 | 3/29 | Klasse I |
| HLA-B | 8/8 | 8/35 | 60/8 | 60/35 | Klasse I |
| HLA-Cw | 7/7 | 7/4 | 3/7 | 3/4 | Klasse I |
| HLA-DRB1* | 13:01/07:01 | 13:01/03:01 | 08:01/07:01 | 08:01/03:01 | Klasse II |
| HLA-DQA1* | 01:03/02:01 | 01:03/05:01 | 04:01/02:01 | 04:01/05:01 | Klasse II |
| HLA-DQB1* | 06:03/03:03 | 06:03/02:01 | 04:02/03:03 | 04:02/02:01 | Klasse II |
| HLA-DPB1* | 04:01/09:01 | 04:01/04:01 | 15:01/09:01 | 15:01/04:01 | Klasse II |
